Münster - Die Zellen von Organismen sind aus Untereinheiten aufgebaut, die sich aus einer Vielzahl einzelner Moleküle zusammensetzen. Wie sich diese unterschiedlichen Proteine auf molekularer Ebene anordnen, ist in vielen Fällen allerdings noch unverstanden, da es an geeigneten Analyseverfahren mangelt.
Forscherinnen und Forscher an der Westfälischen Wilhelms-Universität (WWU) Münster haben nun in Zusammenarbeit mit Kollegen am Max-Planck-Institut für Biochemie, München, ein Verfahren entwickelt, mit dem sich die Anordnung und die Dichte von einzelnen Proteinen in Zellen bestimmen lassen. Durch die Anwendung der neuen Analysemethode wies das Team einen Komplex aus drei sogenannten Adhäsionsproteinen nach, der für die Fähigkeit von Zellen, sich im Gewebe anheften zu können, eine zentrale Rolle zu spielen scheint. Die Studienergebnisse wurden jetzt in der Fachzeitschrift „Nature Communications“ veröffentlicht.
Hintergrund und Methode
Die Anheftung („Adhäsion“) von Zellen wird durch multi-molekulare Adhäsionskomplexe vermittelt, die aus Hunderten von verschiedenen Proteinen aufgebaut sind. Durch die Entwicklung von Superauflösungs-Mikroskopieverfahren, für die 2014 der Nobelpreis verliehen wurde, konnten in den vergangenen Jahren grundlegende Organisationsstrukturen innerhalb dieser Komplexe nachgewiesen werden. Allerdings blieb unklar, wie einzelne Proteine miteinander interagieren und funktionelle Organisationseinheiten bilden. Die Arbeitsgruppen um Prof. Dr. Carsten Grashoff an der WWU Münster am Institut für molekulare Zellbiologie und Prof. Dr. Ralf Jungmann vom Max-Planck-Institut für Biochemie haben nun ein Verfahren entwickelt, mit der die Untersuchung und quantitative Analyse solcher Organisationen selbst in molekular dichten Untereinheiten möglich ist.
„Die wesentliche Limitation selbst der besten hochauflösenden Mikroskopie-Techniken ist, dass nie wirklich alle Moleküle einer Population sichtbar gemacht werden können. Daher war es nahezu unmöglich, qualitative Aussagen zu molekularen Komplexbildungsprozessen zu machen“, erläutert Lisa Fischer, Doktorandin in der AG Grashoff und Erstautorin der Studie. Die neue Methode könne diese Problematik nun umgehen, indem sie experimentelle Untersuchungen mit Computersimulationen kombiniere.
„Durch die Anwendung des neuen Verfahrens konnten wir einen Nachweis für die Existenz eines lang postulierten, aus drei Proteinen bestehenden Adhäsionskomplexes liefern. Wir wussten zwar bereits, dass jedes dieser Moleküle eine wichtige Rolle für die Adhäsion von Zellen spielt. Aber es war nicht klar, dass diese drei Proteine sich auch tatsächlich zu einem Komplex vereinen“, unterstreicht Lisa Fischer. Die Daten zeigten, dass die Kenntnis solcher Prozesse wichtig sei, um die Biologie von Zellen wirklich verstehen zu können. Da die Methode breit anwendbar ist, gehen die Forscher davon aus, dass viele weitere zelluläre Prozesse mit dem neuen Analyseverfahren untersucht werden können.
Finanzierung
Die Arbeiten wurden durch die Deutsche Forschungsgemeinschaft (DFG) gefördert.
Originalpublikation
L.S. Fischer, C. Klingner, T. Schlichthaerle, M.T. Strauss, R. Böttcher, R. Fässler, R. Jungmann, C. Grashoff. Quantitative single-protein imaging reveals molecular complex formation of integrin, talin, and kindlin during cell adhesion. Nature Communications 12, 919 (2021). DOI: 10.1038/s41467-021-21142-2
WWU Münster
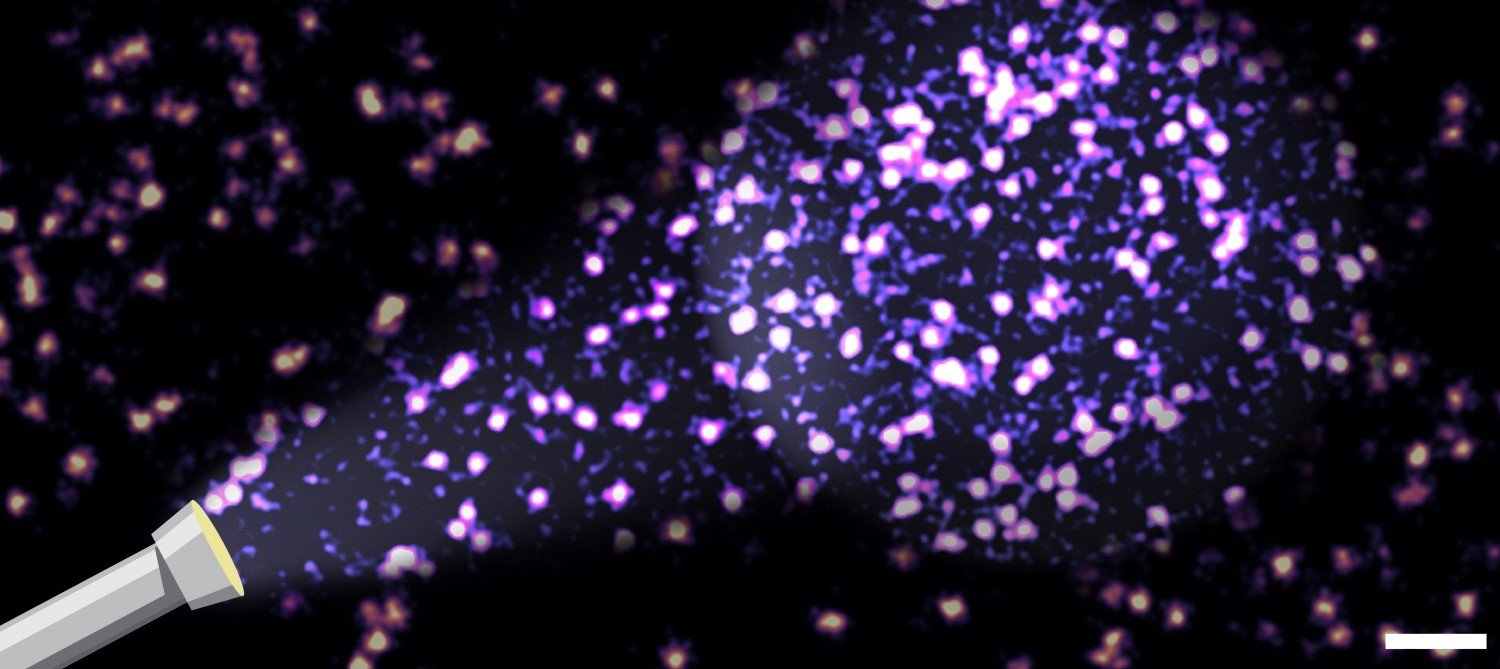
Foto: Lisa Fischer und Carsten Grashoff. Lokalisationssignale einzelner Adhäsionsproteine in Zellen. Bei bisherigen Verfahren blieb ein wesentlicher Anteil der Moleküle unerkannt. Durch das neue Verfahren können die tatsächlich zugrunde liegenden molekularen Parameter bestimmt werden. Maßstabsleiste rechts unten: 100 nm