Wie in einem Orchester interagieren mehrere Helferproteine beim Entstehen der Ribosomen-Bausteine miteinander. Unter ihnen findet sich eines, das den gesamten Prozess anleitet wie ein Dirigent, das Protein ObgE. Dies abzubilden, ist dem Team erstmals gelungen. Die Arbeit ist jetzt im Fachmagazin Molecular Cell erschienen.
Ribosomen sind wesentliche Bestandteile einer jeden Zelle. Häufig
werden sie als Proteinfabriken bezeichnet, denn sie übersetzen
genetische Informationen aus dem Erbgut in Ketten miteinander
verknüpfter Aminosäuren, die Proteine. Auch in Bakterien, wie
beispielsweise dem gut bekannten Darmbakterium Escherichia coli,
findet die Eiweißproduktion innerhalb der Zelle – die
Proteinbiosynthese – auf diese Weise statt. Bleibt der Prozess aus,
stirbt die Zelle.
Einzeller wie E. coli oder andere Bakterien können nicht weiter existieren. Diese Tatsache soll in der Entwicklung von Antibiotika nutzbar gemacht werden. Denn Antibiotika-Resistenzen nehmen zu, neue multiresistente Keime entstehen und verbreiten sich. Gleichzeitig sind seit Langem keine neuen Stoffklassen von Antibiotika entwickelt worden. Ziel neuartiger Ansätze könnte es beispielsweise sein, in den Entstehungsprozess der Ribosomen einzugreifen und ihren Zusammenbau zu blockieren.
„Wir haben es derzeit zufällig mit
einer viralen Pandemie zu tun. Die nächste Pandemie kann durchaus
bakteriellen Ursprungs sein, weil sich Antibiotika-Resistenzen und sogar
multiple Resistenzen rasch über Speziesgrenzen hinweg im Reich der
Bakterien ausbreiten“, erklärt Prof. Dr.
Christian Spahn, Direktor des Instituts für Medizinische Physik und
Biophysik der Charité und Letztautor der aktuellen Studie. „Ziel unserer
Grundlagenforschung ist es daher, langfristig zur Entwicklung neuer
Antibiotika beizutragen.“
Gemeinsam mit Forschenden am Max-Delbrück-Centrum für Molekulare Medizin in der Helmholtz-Gemeinschaft (MDC) und an der Universität Konstanz sind die Charité-Wissenschaftler der Frage nachgegangen, wo genau sich in frühen Prozessen der Ribosomen-Entstehung potentielle Angriffspunkte für neue antibakterielle und antimikrobielle Wirkstoffe finden lassen.
Ribosomen bestehen aus zwei Untereinheiten, einer kleineren und einer größeren. Im Fokus der aktuellen Arbeit des Teams um Dr. Rainer Nikolay, Institut für Medizinische Physik und Biophysik der Charité, stand die große ribosomale Untereinheit des Bakteriums E. coli und ihr Entstehungsprozess. Als mögliches Ziel neuartiger Antibiotika wollten die Wissenschaftlerinnen und Wissenschaftler Vorstufen dieser großen Untereinheit – sogenannte Vorläufer – möglichst nativ, also unverändert, isolieren und abbilden.
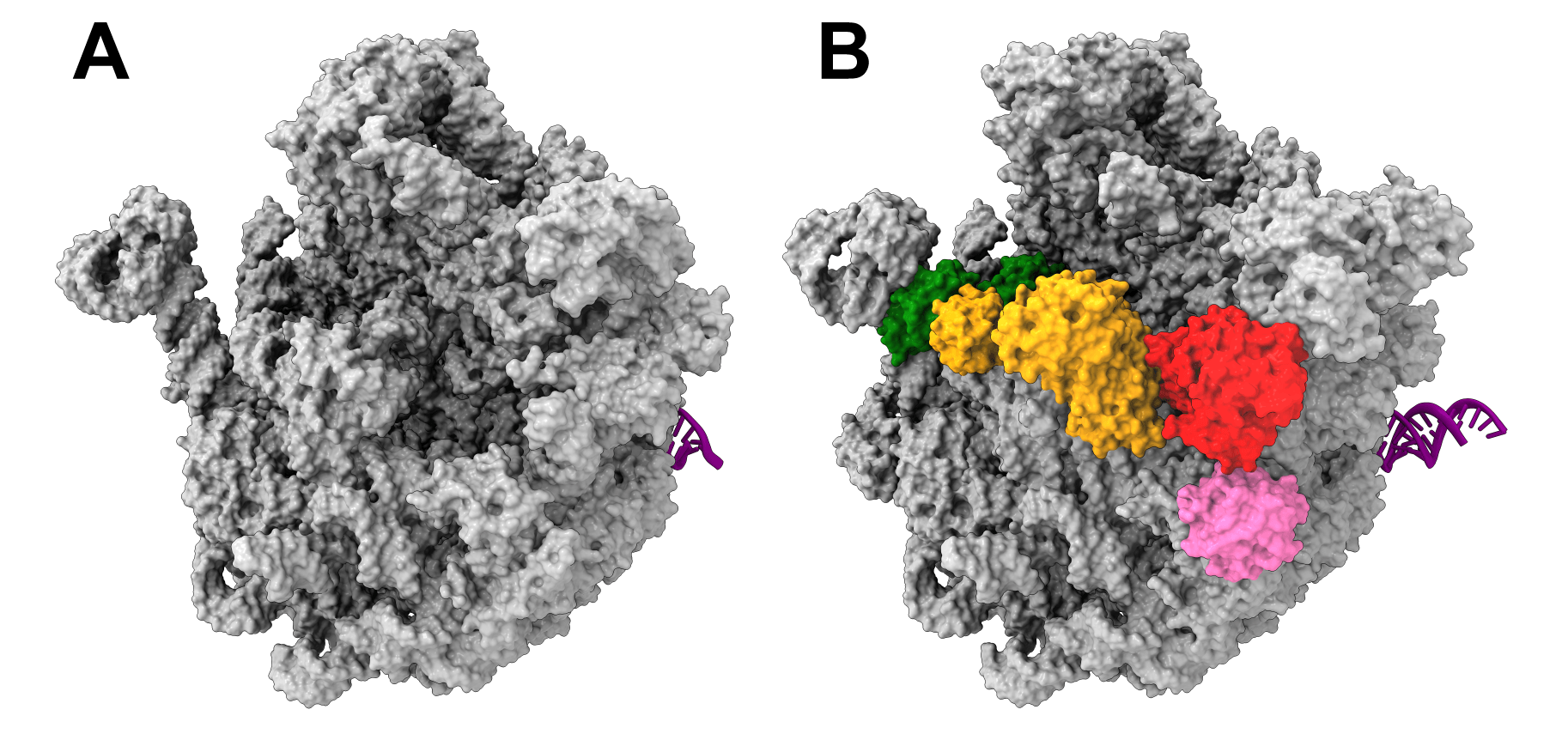
Es ist ihnen erstmals gelungen, einen solchen Vorläufer aus bakteriellen Zellen, in diesem Fall E. coli, zu entnehmen und die Molekülstruktur mithilfe kryo-elektronenmikroskopischer Aufnahmen in nahezu atomarer Auflösung darzustellen. „Wir verstehen nun auf molekularer Ebene besser, wenn auch bei Weitem noch nicht vollständig, wie sich die große ribosomale Untereinheit in einer bakteriellen Zelle formiert“, so Erstautor Dr. Nikolay.
Um die Bakterienzelle für ihre Beobachtungen so wenig
wie möglich zu manipulieren, ist das Forschungsteam quasi minimalinvasiv
vorgegangen. Einem Schlüsselspieler im gesamten Prozess der
Ribosomen-Entstehung, dem Protein ObgE, wurde eine Markierung
angeheftet, ein sogenannter Strep-Tag. Dies geschieht durch einen
Eingriff in das Erbgut des Bakteriums, man bezeichnet den Vorgang als
Knock-in. In der Folge produziert das Bakterium ausschließlich
markiertes ObgE, das nach rascher Aufbereitung der Zellen
elektronenmikroskopisch dargestellt werden kann.
So war es erstmals möglich, den gesamten Komplex zu untersuchen, denn das Helferprotein ObgE hat den Vorläufer der großen ribosomalen Untereinheit sozusagen Huckepack dabei. Mit überraschendem Ergebnis, wie Dr. Nikolay erklärt: „Es stellte sich heraus, dass dieser Vorläufer von zahlreichen Helferproteinen bedeckt ist, die miteinander wechselwirken oder regelrecht kommunizieren. Das Protein ObgE nimmt dabei eine Schlüsselrolle ein, indem es den ganzen Prozess anleitet und orchestriert.“ Genau hier könnten neue Wirkstoffe ansetzen, den Zusammenbau funktionstüchtiger Ribosomen blockieren und somit Bakterienwachstum verhindern.
Mit ähnlichen Strategien will das
Team nun weitere Einblicke in den Entstehungsprozess bakterieller
ribosomaler Untereinheiten erhalten und die molekularbiologischen
Abläufe noch besser verstehen. Bereits abgeschlossene Arbeiten an der
Charité und am Max-Planck-Institut für molekulare Genetik hatten
wertvolle Informationen über den grundsätzlichen Aufbau und
unterschiedliche Reifegrade der zellulären Proteinfabriken geliefert.
Allerdings basierten diese Einblicke bislang auf Studien im Reagenzglas, während nun die Bildung der großen ribosomalen Untereinheit in einer lebenden Zelle nachvollzogen werden konnte. Dieser Schritt ist entscheidend, denn um zelluläre Angriffspunkte gänzlich neuer pharmazeutischer Stoffe zu ermitteln, müssen die Forschenden Unterschiede im Prozess der Ribosomen-Entstehung in Bakterien und in menschlichen Zellen ausfindig machen.
„Dem sind wir jetzt ein Stück nähergekommen“, so Dr. Nikolay. „Wir konnten sowohl konservierte als auch sich unterscheidende evolutionäre Merkmale zwischen Prokaryoten – wie es Bakterien sind – und Eukaryoten – Lebewesen, bei denen das Erbgut in Zellkernen vorliegt – aufzeigen.“ Diese Erkenntnisse sind wichtig, um bakterienspezifische Merkmale ins Visier zu nehmen und gleichzeitig menschliche Zellen vor unerwünschten Nebenwirkungen zu schützen.
Über die Studie
Unterstützt wurden die Arbeiten durch die Konstanz Research School Chemical Biology, die Human Frontier Science Program Organization, die Deutsche Forschungsgemeinschaft (DFG) Grants FOR1805 und SFB740, die Freie Universität Berlin und das Land Berlin.
Charité Universitätsmedizin Berlin
Graphik: Nikolay/Charité. 3D-Strukturmodelle von Vorläufern der großen bakteriellen ribosomalen Untereinheit. A) Im Reagenzglas (in vitro) erzeugter Vorläufer-Partikel. B) Der Zelle entnommener Vorläufer-Partikel, der charakteristische Anzeichen seiner natürlichen Herkunft aufweist. Markiert sind die gebundenen Helferproteine: YjgA (grün), RluD (orange), ObgE (rot) und RsfS (pink).